GenBank
GenBank és una base de dades pública que conté seqüències de nucleòtids i anotacions bibliogràfiques i biològiques de suport. Va ser creada pel Laboratori Nacional Los Álamos i és distribuïda pel Centre Nacional per la Informació de Biotecnologia (NCBI), una divisió de la Biblioteca Nacional de Medicina dels Estats Units (NLM). GenBank forma part de la Col·laboració Internacional de Bases de Dades de Seqüències de Nucleòtids (International Nucleotide Sequence Database Collaboration, INSDC) i intercanvia dades amb l'Arxiu Europeu de Nucleòtids (ENA) i el DNA DataBank del Japó (DDBJ) diàriament.[1]
 | |
| URL | https://www.ncbi.nlm.nih.gov/genbank/ |
|---|---|
| Tipus | Base de dades de seqüències de nucleòtids |
| Tema | medicina i gen |
| Llicència | Domini públic - Govern dels Estats Units |
| Part de | Centre Nacional per la Informació de Biotecnologia - NCBI |
| Creador | Laboratori Nacional Los Álamos |
| Llançament | 1979 |
| Seu | Bethesda, Maryland |
| Estat | Actiu |
L’objectiu de Genbank és garantir que hi hagi una col·lecció uniforme i completa d’informació de seqüències a tot el món. L’NCBI posa a disposició les dades de GenBank sense cap cost a través d’una àmplia gama de serveis de recuperació, entre ells Internet i FTP.[2]
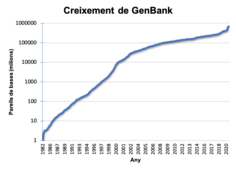
NCBI construeix GenBank principalment a partir d’enviaments de dades de seqüències de laboratoris individuals, a més de contribucions massives de centres de seqüenciació de gran escala. GenBank i els seus col·laboradors reben seqüències de més de 100,000 organismes diferents produïdes a laboratoris arreu del món.[1]
La base de dades va ser creada el 1979 en el Laboratori Nacional de Los Alamos (LANL), a Nou Mèxic, EEUU, per Walter Goad. GenBank ha esdevingut una base de dades important per a la recerca en camps biològics i en els darrers anys ha crescut a un índex exponencial, doblant el número de dades cada 12 mesos.[1]
La versió 240, publicada a l’octubre del 2020, conté més de 650 mil milions de parells de bases, en més de 200 milions de seqüències. GenBank és construït amb contribucions de laboratoris individuals, així com d’enviaments massius de centres de seqüenciació a gran escala.[3]
Les seqüències obtingudes de GenBank es poden utilitzar per fer estudis d'alineament de seqüències mitjançant el programa informàtic BLAST (Basic Local Alignment Search Tool).[4]
Història
modificaGenBank es va crear el 1979 al Laboratori Nacional Los Alamos i es va anomenar Los Alamos Sequence Database. El projecte va ser impulsat per Walter Goad, físic nuclear del grup de Biologia i Biofísica Teòrica, i el seu equip amb l’objectiu de crear un projecte pilot per a recollir seqüències de nucleòtids que es podrien utilitzar per a l'anàlisi.[5][6]
El 1982 Los Alamos Sequence Database va prendre el nom de GenBank i es va convertir en una base de dades pública. Aquest fet coincideix amb l’adjudicament d’un contracte de dos anys per 5 milions de dòlars, per establir i operar la base de dades al LANL.[5] El finançament va ser proporcionat pels Instituts Nacionals de Salut (NIH), la Fundació Nacional de Ciències (NSF), el Departament d'Energia (DOE), i el Departament de Defensa (DOD).[7]
A més, el projecte GenBank va iniciar grups de notícies BIOSCI/Bionet per promoure comunicacions d'accés obert entre biocientífics.[7]A mitjans dels anys vuitanta, el projecte va ser gestionat juntament amb Intelligenetics, empresa de la Universitat Stanford. Aquest va ser un dels primers projectes comunitaris de bioinformàtica a Internet, amb la finalitat d’aconseguir comunicacions lliures entre els científics.[5]Entre el 1989 i el 1992, GenBank va passar a la recentment creada NCBI, una divisió de la Biblioteca Nacional de Medicina (NLM), situada al campus dels Instituts Nacionals de Salut dels Estats Units (Bethesda, Maryland).L’any 1993 l’NCBI comença a acceptar presentacions directes a GenBank.[5]
Contribucions
modificaAl GenBank només es poden entregar seqüències originals. Els investigadors envien les dades a GenBank o a les bases de dades col·laboradores (ENA i DDBJ). Les dades s’intercanvien diàriament entre aquestes tres bases de dades, de manera que les actualitzacions diàries dels servidors NCBI incorporen les dades de seqüència més recentment disponibles de totes les fonts.[8]
Les contribucions poden ser enviades de manera electrònica. Moltes revistes científiques requereixen que els autors amb dades de seqüència les enviïn a una base de dades de seqüències pública com a condició de publicació. Les dades de seqüència es poden enviar a través de diferents portals:[8][9]
- BankIt. Mitjançant aquesta eina es poden enviar tant conjunts de seqüències com seqüències úniques. Aproximadament un terç de les presentacions dels autors es reben a través d'aquesta eina de presentació de dades. Amb BankIt, els autors ingressen la informació de la seva seqüència i les pertinents anotacions biològiques en un formulari que permet el remitent descriure la seqüència més detalladament sense haver d'aprendre regles de format.
- Portal d’enviament de l’NCBI. És un sistema centralitzat on es poden enviar genomes procariotes i eucariotes. Aquest portal proporciona assistents per ajudar els casos d’enviament habituals.
- t2blastn. Permet als remitents convertir una taula d’anotacions generades a partir d’una canalització d’anotacions en un registre adequat per enviar-lo a GenBank. Aquests fitxers per a enviaments arriben a GenBank a través del portal de presentació.
- Genome Workbench. Ofereix un gran conjunt d'eines integrades per estudiar i analitzar dades genètiques, explorar-les i comparar-les amb dades de diverses fonts.
Organització de la base de dades
modificaDivisions
modificaGenBank assigna cada registre de seqüència a una divisió segons la taxonomia d’origen o l'estratègia de seqüenciació utilitzada per obtenir les dades. Hi ha dotze divisions taxonòmiques (BCT, ENV, INV, MAM, PHG, PLN, PRI, ROD, SYN, UNA, VRL, VRT) i cinc divisions d’alt rendiment (EST, GSS, HTC, HTG, STS).[1][10]
A part, trobem altres tipus de divisions classificades individualment: [10]
- La divisió PAT (Patent Sequences) conté registres subministrats per les oficines de patents.
- La divisió TSA (Transcriptome Shotgun Assembly) conté seqüències obtingudes a partir de la seqüenciació “per perdigonada” (shotgun) de transcriptomes.
- La divisió WGS (Whole Genome Shotgun data) conté seqüències obtingudes a partir de la seqüenciació “per perdigonada” (shotgun) del genoma complet.
- La divisió TLS (Targeted Locus Studies) conté conjunts de seqüències de rRNA 16S o elements ultraconservats (UCEs).
La mida i el creixement d’aquestes divisions (en parells de bases) i de GenBank en general es mostren a la següent taula.
| Divisions | Descripció | Versió 233 (agost 2019) | Increment anual (%)a |
|---|---|---|---|
| SYN | Sintètiques | 7 701 613 755 | 545.96% |
| VRT | Altres vertebrats | 46 205 911 214 | 342.51% |
| PLN | Plantes | 59 248 524 178 | 157.29% |
| UNA | Sense anotacions | 548 041 | 84.71% |
| WGS | Dades de genomes complets | 5 585 922 333 160 | 74.30% |
| TLS | Estudis de locus diana | 10 531 800 829 | 73.28% |
| INV | Invertebrats | 12 578 394 104 | 46.31% |
| PHG | Bacteriòfags | 637 015 044 | 37.58% |
| BCT | Bacteris | 72 495 994 966 | 35.40% |
| TSA | Dades de seqüenciació de transcriptomes | 294 727 165 179 | 30.69% |
| VRL | Virus | 4 782 719 535 | 17.40% |
| PAT | Seqüències de patents | 24 715 727 030 | 12.24% |
| ENV | Mostres mediambientals | 6 139 560 312 | 5.51% |
| PRI | Primats | 8 491 950 612 | 2.78% |
| HTC | cDNA d'alt rendiment | 728 868 423 | 1.03% |
| MAM | Altres mamífers | 6 258 926 080 | 0.71% |
| EST | Marcadors de seqüència expressada | 43 280 039 563 | 0.68% |
| ROD | Rosegadors | 4 554 525 905 | 0.43% |
| HTG | Genòmica d'alt rendiment | 27 774 725 922 | 0.01% |
| STS | Llocs marcats per una seqüència | 640 918 572 | 0.01% |
| GSS | Seqüències d'estudi del genoma | 26 339 260 641 | 0.00% |
| TOTAL | Totes les seqüències de GenBank | 6 233 224 722 236 | 69.52% |
aMesura relativa a la versió 227 (agost 2018).
Respecte els notables augments de l’últim any, a la taula s’inclouen la presentació de les 57 construccions cromosòmiques sintètiques i la presentació d’unes 60 seqüències eucariotes a escala cromosòmica de la divisió VRT.[1]
Taxonomia basada en seqüències
modificaLes seqüències de la base de dades es classifiquen i es poden consultar mitjançant una taxonomia basada en seqüències desenvolupada pel NCBI. GenBank compta amb 420.000 espècies descrites. Es mostren el nombre de parells de bases de les principals espècies (sense incloure les de les divisions WGS i TSA) a la següent taula:[10]
| Organisme | Parells de basesb |
|---|---|
| Homo sapiens | 19 752 523 722 |
| Mus musculus | 10 246 475 076 |
| Rattus norvegicus | 6 530 046 440 |
| Bos taurus | 5 431 692 037 |
| Zea mays | 5 245 788 885 |
| Sus scrofa | 5 075 446 882 |
| Hordeum vulgare | 3 237 283 130 |
| Escherichia coli | 3 220 757 391 |
| Danio rerio | 3 191 415 637 |
| Oryzias latipes | 2 836 938 628 |
| Arachis hypogaea | 2 682 391 941 |
| Triticum aestivum | 2 636 490 116 |
| Ovis canadensis | 2 590 574 434 |
| Solanum lycopersicum | 2 572 291 998 |
| Bos mutus | 2 290 216 303 |
| Cyprinus carpio | 1 836 731 087 |
| Oryza sativa | 1 727 115 789 |
| Apteryx australis | 1 595 510 956 |
| Bordetella pertussis | 1 456 386 736 |
| Strongylocentrotus purpuratus | 1 436 247 256 |
bVersió 227 (agost 2018). Exclou seqüències de cloroplasts, mitocondris, metagenomes, organismes no cultivats, WGS, TSA i la divisió CON.
Registres
modificaLa informació de cada seqüència està continguda en un registre de GenBank, accessible mitjançant el cercador de la pàgina web de GenBank. Cada registre per a cada seqüència conté, entre d’altres, la seqüència, el locus, el número d’accés, la versió, la descripció, els comentaris, els autors i l’organisme i taxonomia.[11] Genbank ofereix un exemple de registre a la seva pàgina web.
Identificador de seqüència
modificaUn cop enviada la seqüència, a cada registre GenBank se li assigna un identificador únic anomenat número d’accés. Aquest número és compartit amb les tres bases de dades col·laboradores (GenBank, DDBJ, ENA). El número d’accés es mantindrà igual encara que hi hagi un canvi en la seqüència o l’anotació. Quan hi ha canvis en les dades de la seqüència s’identifica com una nova versió d’aquesta, afegint el número de la versió darrere del número d’accés.[10]Existeix un informe de l’historial de revisions que resumeix les diverses actualitzacions d’un registre determinat, incloent tots els canvis, també els que no són de seqüència.[7]
Els identificadors de seqüències WGS, TSA i TLS funcionen diferent. Aquestes dades són enviades com a part de grans grups de projectes, i cada projecte compta amb un registre que agrupa enllaços a les seqüències individuals. Cada seqüència individual dins un projecte tindrà un número d’accés que comença amb l’identificador del projecte (número d’accés principal).[7]
El número d’accés d’una seqüència amb la versió corresponent, a més de ser l’identificador principal d’un registre de seqüències GenBank, també és la forma més eficient i fiable de citar un registre de seqüències en publicacions. La cerca amb un número d’accés a GenBank (sense el sufix de versió) recuperarà la versió més recent d’un registre, que pot haver canviat des de la publicació del document.[7]
Procés de verificació de genomes i publicació
modificaDe mitjana, el personal de GenBank triga dos dies a assignar un número d’accés a un enviament de seqüències. No obstant, aquest temps pot variar en funció de la complexitat de l'enviament, els genomes complets solen requerir més temps. El personal de GenBank assigna aproximadament 3.500 accessos al dia. Els enviaments directes reben una revisió de garantia de qualitat. En la revisió es fa èmfasi en comprovacions de la contaminació de vectors genètics, la traducció adequada de les regions de codificació, la taxonomia correcta i les citacions bibliogràfiques correctes. L’autor rep un esborrany del registre GenBank perquè el revisi abans que entri a la base de dades. Els autors poden sol·licitar que les seqüències es mantinguin confidencials fins al moment de la publicació. Les contribucions són llavors publicades a la base de dades pública, on les entrades estan disponibles via Entrez i es poden descarregar per FTP.[1]
Seqüències no verificades
modificaEn el cas de que el personal de Genbank no pugui confirmar la precisió de les dades o anotacions de la seqüència presentada durant el procés de revisió estàndard, aquesta seqüència es podrà etiquetar com a no verificada.[1] Fins que l'emissor no pugui resoldre aquests problemes, al registre apareixerà la seqüència com a no verificada i s’inclourà una breu descripció dels problemes. A més, les seqüències no verificades no s’inclouran a les bases de dades de BLAST.[10]
Informació addicional
modificaReferències
modifica- ↑ 1,0 1,1 1,2 1,3 1,4 1,5 1,6 1,7 Sayers, Eric W.; Cavanaugh, Mark; Clark, Karen; Ostell, James; Pruitt, Kim D. «GenBank». Nucleic Acids Research, 48, D1, 01-08-2020, pàg. D84–D86. DOI: 10.1093/nar/gkz956. ISSN: 1362-4962. PMC: 7145611. PMID: 31665464.
- ↑ Benson, Dennis A.; Cavanaugh, Mark; Clark, Karen; Karsch-Mizrachi, Ilene; Ostell, James «GenBank». Nucleic Acids Research, 46, D1, 01-04-2018, pàg. D41–D47. DOI: 10.1093/nar/gkx1094. ISSN: 1362-4962. PMC: 5753231. PMID: 29140468.
- ↑ «Current GenBank Release Notes» (en anglès). [Consulta: 19 desembre 2020].
- ↑ «GenBank Overview». [Consulta: 20 desembre 2020].
- ↑ 5,0 5,1 5,2 5,3 «DOE Genome Informatics -- Announcement II». [Consulta: 19 desembre 2020].
- ↑ Choudhuri, Supratim. Bioinformatics for Begginers, p. 80. ISBN 978-0-12-410471-6.
- ↑ 7,0 7,1 7,2 7,3 7,4 Benton, David «Recent changes in the GenBank ® On-line Service» (en anglès). Nucleic Acids Research, 18, 6, 1990, pàg. 1517–1520. DOI: 10.1093/nar/18.6.1517. ISSN: 0305-1048. PMC: PMC330520. PMID: 2326192.
- ↑ 8,0 8,1 «How to submit data to GenBank». [Consulta: 19 desembre 2020].
- ↑ «What is tbl2asn?». [Consulta: 19 desembre 2020].
- ↑ 10,0 10,1 10,2 10,3 10,4 10,5 Sayers, Eric W.; Cavanaugh, Mark; Clark, Karen; Ostell, James; Pruitt, Kim D. «GenBank». Nucleic Acids Research, 47, D1, 01-08-2019, pàg. D94–D99. DOI: 10.1093/nar/gky989. ISSN: 1362-4962. PMC: 6323954. PMID: 30365038.
- ↑ «Sample GenBank Record». [Consulta: 19 desembre 2020].